FA_DS08_1e
Hayashi Atsuhiro
2020/11/10
1. 先週のショート課題から: 11名
- 散布図の利用について。それぞれのサンプルがどこに布置しているかを見ることができる。その軸の特徴をより強く持ったサンプルが外側に布置されることになる。また、似た傾向のサンプルが近い距離に布置されることから、グルーピングにも用いられる。
- 残差の値について。「残差が20以上」と紹介したのは一例であり、毎回「20」である必要はありません。0近傍に均等に分布していることが期待されます。
2. 因子分析
いくつか(p個)の変量の情報を、損失をできるだけ少なくして、 少数変量(m個、m<p)の総合的指標(主成分)で代表させる方法として 主成分分析(Principal Component Analysis, PCA)と 因子分析(Factor Analysis, FA)がある。 テストの成績を総合した総合的成績、 いろいろな症状を総合した総合的な重症度、 種々の財務指標に基づく企業の評価 等を求めたいといった場合に用いられる。 p変量(p次元)の観測値をm個(m次元)の成分に縮約させるという意味で、 次元を減少させる(reduce)方法と言うこともでき、 多変量データを要約する際の有力な方法である。
両者は似た目的に使われるが、元になっている考え方は異なるので 利用する場面では注意が必要である。 違いに焦点を当てながら前回の主成分分析に続いて今回は因子分析を紹介する。
前述の主成分分析の場合は、 データの散らばり方(分散)を捉えてデータ特性を把握する手法であった。 一方、因子分析は、変数間に(潜在的な)構造を持ち込んで関係を探る手法である (少し理解しにくいかもしれないが)。 この手法は心理学の分野で広く利用されている。
-
定式化
-
例えば、i番目の学生の3科目の試験成績
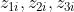 が得られていたとする(i=1,2,…,n)。 fiをi番目の学生の特徴を表す要素、ajをj番目の科目の特徴を表す要素とし、 それらが以下のような積の形で表現できたとしたら、学生個人と科目の特性が分離できることになる。
が得られていたとする(i=1,2,…,n)。 fiをi番目の学生の特徴を表す要素、ajをj番目の科目の特徴を表す要素とし、 それらが以下のような積の形で表現できたとしたら、学生個人と科目の特性が分離できることになる。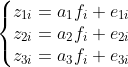
-
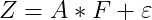
-
ここで、
-
測定対象
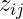 : 成績、測定値、…。
: 成績、測定値、…。
-
共通因子(潜在変量) :
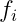 : 因子得点(測定不能)、個体の特徴付け、散布図に、i=1,2,…,n.
: 因子得点(測定不能)、個体の特徴付け、散布図に、i=1,2,…,n.
-
因子負荷量(潜在変量) :
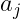 : 因子の特徴付け、j=1,2,…,p.
: 因子の特徴付け、j=1,2,…,p.
-
独自因子 :
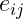 : 変動、誤差
: 変動、誤差
-
測定対象
-
いくつかの仮定 :
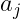 ,
, 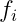 ,
, 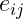 の下で、これらを満たす
の下で、これらを満たす 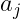 ,
, 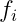 を求める。
を求める。
-
回転の不定性:
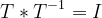 となる回転行列Tを用いると、
となる回転行列Tを用いると、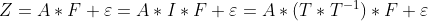
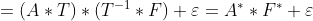
となり、AとFの対は無限組存在する。一意には決まらない。 何らかの基準を条件に確定させる。
-
-
p次元を何次元に縮約するかの軸数(因子数)も利用者側から与える必要がある。
- 因子の解釈
- 因子軸の回転 : 直交回転、斜交回転
- 因子数を指定して分析を進める必要があるため、 前処理や確定した因子数に基づく分析等、 行きつ戻りつの試行錯誤が必要な手法である。
3. [例題] 食品の嗜好性を探ってみよう
主成分分析の時も利用した100種類の食品の性、年齢毎の嗜好度調査のデータを例に。
-
まずは因子数を決めよう :
-
解釈方法 :
-
固有値(Eigenvalue) : 相関行列の固有値の振る舞いから因子数(軸数)を決める。
-
因子数の決定 : 解析者側の判断
-
固有値の大小、減少傾向から因子数を決める。
- 固有値の変化量からすると、3 でも良さそう : 3 と 4 の間が空いてる
- 因子数を 3 として計算してみよう
-
固有値の大小、減少傾向から因子数を決める。
-
因子数の決め方は、主成分分析の時と同様の考え方
- 累積寄与率(Cumulative)
- 固有値の値(Eigenvalue, Proportion)
- 固有値間のギャップ(Difference) 等
# データの読み込み 100品目の食品データ food<-read.csv("food.csv", skip=17, header=TRUE, na.strings="NA") dim(food)## [1] 100 10colnames(food)## [1] "Group01" "Group02" "Group03" "Group04" "Group05" "Group06" "Group07" ## [8] "Group08" "Group09" "Group10"food[1:5,]## Group01 Group02 Group03 Group04 Group05 Group06 Group07 Group08 Group09 ## 1 7.69 7.31 7.47 7.76 7.87 7.51 7.24 7.70 7.91 ## 2 6.59 5.56 6.21 6.04 5.81 6.64 6.11 6.53 6.44 ## 3 4.55 4.18 4.36 4.25 4.53 4.60 3.66 4.04 3.68 ## 4 6.78 6.11 6.30 5.98 5.56 6.37 6.29 5.43 5.32 ## 5 6.47 6.24 6.02 5.42 5.88 6.00 5.60 4.60 5.40 ## Group10 ## 1 7.95 ## 2 6.64 ## 3 4.43 ## 4 5.28 ## 5 5.95corrFood = cor(food, use="complete.obs" ) # 相関行列を計算 corrFood## Group01 Group02 Group03 Group04 Group05 Group06 Group07 ## Group01 1.0000000 0.8707502 0.5158024 0.3701171 0.1722555 0.9383574 0.8106677 ## Group02 0.8707502 1.0000000 0.7588002 0.6042866 0.4021423 0.8206969 0.8380528 ## Group03 0.5158024 0.7588002 1.0000000 0.8524283 0.7261755 0.5164491 0.6583837 ## Group04 0.3701171 0.6042866 0.8524283 1.0000000 0.8742306 0.3580258 0.4875341 ## Group05 0.1722555 0.4021423 0.7261755 0.8742306 1.0000000 0.2077093 0.3543237 ## Group06 0.9383574 0.8206969 0.5164491 0.3580258 0.2077093 1.0000000 0.8887956 ## Group07 0.8106677 0.8380528 0.6583837 0.4875341 0.3543237 0.8887956 1.0000000 ## Group08 0.6160570 0.7095440 0.6989555 0.6198739 0.5234535 0.7464624 0.8949360 ## Group09 0.5003457 0.6469773 0.7013245 0.7206826 0.7100951 0.6214719 0.7678802 ## Group10 0.3298161 0.4569242 0.5584082 0.6321387 0.7479457 0.4931943 0.6415215 ## Group08 Group09 Group10 ## Group01 0.6160570 0.5003457 0.3298161 ## Group02 0.7095440 0.6469773 0.4569242 ## Group03 0.6989555 0.7013245 0.5584082 ## Group04 0.6198739 0.7206826 0.6321387 ## Group05 0.5234535 0.7100951 0.7479457 ## Group06 0.7464624 0.6214719 0.4931943 ## Group07 0.8949360 0.7678802 0.6415215 ## Group08 1.0000000 0.8528403 0.7741073 ## Group09 0.8528403 1.0000000 0.9111652 ## Group10 0.7741073 0.9111652 1.0000000eigenFood<-eigen(corrFood)$values # 相関行列の固有値を計算 eigenFood## [1] 6.82795512 1.76187311 0.75445124 0.26237637 0.12155202 0.09796547 ## [7] 0.07209967 0.04408041 0.03575249 0.02189408# 固有値の減少傾向を視覚的に捉えるためためにプロットしてみる plot(eigenFood, type="b", main="Scree Plot", xlab="NUM", ylab="Eigen Value") abline(h=1, lty=3) # y = 1 の横線を追加 -
固有値(Eigenvalue) : 相関行列の固有値の振る舞いから因子数(軸数)を決める。
4. 因子数を3に指定して解析してみる
# 因子分析1
FAresultFood<-factanal(x=food, factors=3,
rotation="none", scores="Bartlett") # scores="regression")
# 分析結果要約
print(FAresultFood, cutoff=0)##
## Call:
## factanal(x = food, factors = 3, scores = "Bartlett", rotation = "none")
##
## Uniquenesses:
## Group01 Group02 Group03 Group04 Group05 Group06 Group07 Group08 Group09 Group10
## 0.055 0.115 0.158 0.059 0.108 0.035 0.113 0.183 0.083 0.025
##
## Loadings:
## Factor1 Factor2 Factor3
## Group01 0.735 0.629 0.097
## Group02 0.818 0.370 0.279
## Group03 0.773 -0.109 0.482
## Group04 0.737 -0.359 0.519
## Group05 0.672 -0.589 0.305
## Group06 0.817 0.535 -0.105
## Group07 0.888 0.301 -0.091
## Group08 0.899 0.008 -0.091
## Group09 0.919 -0.249 -0.096
## Group10 0.846 -0.427 -0.276
##
## Factor1 Factor2 Factor3
## SS loadings 6.627 1.643 0.795
## Proportion Var 0.663 0.164 0.080
## Cumulative Var 0.663 0.827 0.907
##
## Test of the hypothesis that 3 factors are sufficient.
## The chi square statistic is 158.68 on 18 degrees of freedom.
## The p-value is 1.51e-24- 寄与率(Proportion Var) : 各因子の説明力の大きさ
- 累積寄与率(Cumulative Var) : 3因子まとめての説明力の大きさ : 90.7%
-
因子負荷量(Loadings) :
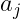 : 大小、符号から因子の特徴を見出す
: 大小、符号から因子の特徴を見出す
- 第1因子 : 全体的な嗜好
- 第2因子 : 年齢効果 (+ 若年、- 年輩)
- 第3因子 : 性別効果 (+ 男性、- 女性)
-
各個体の因子得点(
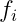 )の散布図 :
)の散布図 :
- 各個体の具体的な位置を把握
- 第2因子と第3因子の関係が面白そう
# 各サンプルの変換点(因子得点, scores)を10ケース出力してみる。
FAresultFood$scores[1:10,]## Factor1 Factor2 Factor3
## [1,] 2.2741629 -0.91142227 0.5617334
## [2,] 0.7767690 -0.23466632 -0.8200170
## [3,] -1.5356049 0.09256539 -1.0014913
## [4,] 0.2122250 0.61886083 0.5953158
## [5,] 0.1782223 0.07551002 -0.3290809
## [6,] 1.4650663 -0.33139282 -0.6440971
## [7,] -0.1573747 0.59937087 -0.2800068
## [8,] 0.2650604 1.35136096 -0.4696926
## [9,] -0.9471523 1.31445858 0.4812331
## [10,] 0.7549617 -0.26779514 -0.2964663# 番号で描画してみる。
plot(FAresultFood$scores[,1], FAresultFood$scores[,2], type="n") # マークを描かない
text(FAresultFood$scores[,1], FAresultFood$scores[,2], cex=.75) # その場所に番号を表示
abline(h=seq(-2,3,1), lty=3)
abline(v=seq(-3,2,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)plot(FAresultFood$scores[,2], FAresultFood$scores[,3], type="n") # マークを描かない
text(FAresultFood$scores[,2], FAresultFood$scores[,3], cex=.75) # その場所に番号を表示
abline(h=seq(-2,4,1), lty=3)
abline(v=seq(-2,3,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)plot(FAresultFood$scores[,3], FAresultFood$scores[,1], type="n") # マークを描かない
text(FAresultFood$scores[,3], FAresultFood$scores[,1], cex=.75) # その場所に番号を表示
abline(h=seq(-3,2,1), lty=3)
abline(v=seq(-2,4,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)5. 回転させてみよう: ただし、回転が必須ではない
- 代表的な直交回転のVarimaxを適用してみる
# 因子分析2
FAresultFood<-factanal(x=food, factors=3,
rotation="varimax", scores="Bartlett")
# 分析結果要約
print(FAresultFood, cutoff=0)##
## Call:
## factanal(x = food, factors = 3, scores = "Bartlett", rotation = "varimax")
##
## Uniquenesses:
## Group01 Group02 Group03 Group04 Group05 Group06 Group07 Group08 Group09 Group10
## 0.055 0.115 0.158 0.059 0.108 0.035 0.113 0.183 0.083 0.025
##
## Loadings:
## Factor1 Factor2 Factor3
## Group01 0.958 0.144 0.080
## Group02 0.822 0.435 0.140
## Group03 0.435 0.779 0.214
## Group04 0.223 0.899 0.289
## Group05 -0.003 0.803 0.496
## Group06 0.932 0.067 0.303
## Group07 0.800 0.211 0.450
## Group08 0.584 0.345 0.598
## Group09 0.401 0.461 0.737
## Group10 0.211 0.365 0.893
##
## Factor1 Factor2 Factor3
## SS loadings 3.888 2.784 2.393
## Proportion Var 0.389 0.278 0.239
## Cumulative Var 0.389 0.667 0.907
##
## Test of the hypothesis that 3 factors are sufficient.
## The chi square statistic is 158.68 on 18 degrees of freedom.
## The p-value is 1.51e-24- 寄与率(Proportion Var) : 各因子の説明力の大きさ
- 累積寄与率(Cumulative Var) : 3因子まとめての説明力の大きさ : 90.7%
-
因子負荷量(Loadings) :
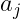 : 大小、符号から因子の特徴を見出す
: 大小、符号から因子の特徴を見出す
- 第1因子 : 若年層の嗜好 (+ 若年、- 年輩)
- 第2因子 : 成人男性の嗜好 (+ 成年男子)
- 第3因子 : 成人女性の嗜好 (+ 成年女子)
-
各個体の因子得点(
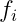 )の散布図 :
)の散布図 :
- 各個体の具体的な位置を把握
- 各因子間の関係が面白そう。ただ、少し難解か?
- 回転前と回転後でどのように解釈が変化したか?
# 番号で描画してみる。
plot(FAresultFood$scores[,1], FAresultFood$scores[,2], type="n") # マークを描かない
text(FAresultFood$scores[,1], FAresultFood$scores[,2], cex=.75) # その場所に番号を表示
abline(h=seq(-2,3,1), lty=3)
abline(v=seq(-2,2,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)plot(FAresultFood$scores[,2], FAresultFood$scores[,3], type="n") # マークを描かない
text(FAresultFood$scores[,2], FAresultFood$scores[,3], cex=.75) # その場所に番号を表示
abline(h=seq(-3,1,1), lty=3)
abline(v=seq(-2,3,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)plot(FAresultFood$scores[,3], FAresultFood$scores[,1], type="n") # マークを描かない
text(FAresultFood$scores[,3], FAresultFood$scores[,1], cex=.75) # その場所に番号を表示
abline(h=seq(-2,2,1), lty=3)
abline(v=seq(-3,1,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)6. 次の一手
- p次元を何次元に縮約するかの因子数は利用者側から与える必要がある。 ===> 上記3因子で解釈に無理はないか?
- 近傍の因子数(2とか4とか)で試してみるのも一案である。
-
回転させないときと、回転させたときでどちらが解釈が容易か?
- 因子数を指定して分析を進める必要があるため、 前処理や確定した因子数に基づく分析等、 行きつ戻りつの試行錯誤が必要な手法である。
- データのバックグラウンドを把握していると、因子の解釈を行い易い。
7. 主成分分析(PCA)と因子分析(FA): 目的は同じでも異なる手法
-
目的: いくつか(p個)の変量の情報を、損失をできるだけ少なくして、 少数変量(m個、m<p)の総合的指標(主成分)で代表させたい。
-
考え方
- 主成分分析 : 分散最大
- 因子分析 : モデルの導入、回転性、一意性に疑問(特に斜交回転)
-
対象データ
- サンプルサイズのある程度大きいデータが対象となる
- 変量数もある程度大きいデータが対象となる
- 当然ながらサンプルサイズの方が変量数よりも大きいこと : 多変量解析全般
- 対象データを熟知している方が解釈しやすい(熟知の必要性)
- 因子の特徴付けはデータのバックグラウンドに深く関係
- 経験を積むとより納得する説明ができる
-
潜在的な構造が仮定できるか? モデルが適用可能な問題か吟味する必要性。
-
軸数、因子数を変えて解釈を行ってみる。行きつ戻りつして試行錯誤してみる。
[ノウハウ] ラインマーカーで絶対値の大きい変量にマークを付けると把握し易くなるのではないか。
- いろいろなデータで経験を積んでみる。
- どっちを使う? : やってみる。解釈してみる。今までの事例と比較してみる。
- …
- データによっては解釈が困難なことも有り得る。 また、自分の思い付かない結果を含んでいることもある。
8. “データサイエンス”に求められるもの
私の担当部分を終えるにあたって、これまでの経験から データサイエンスに付いての若干の私見を述べる。-
ビッグデータ: 明確な定義はない
- 3V: Volume(容量), Velocity(更新頻度), Variety(多様性)
-
AI ブーム: 過去の蓄積から似たデータを取り出す
- データストレージの発達
-
センサーの充実、普及 ===> 大量データ の生成時代
- 「押し寄せてくるデータ」への対処
-
【これまで】 能動的なデータ採取 ===> 【これから】 受動的なデータ採取
-
初等中等教育にも「統計教育」が導入。大学入試には H27年度から。
- 思考力、判断力、表現力、読解力
- 「統計的なものの見方や考え方」を身に付けてもらう
- 知識暗記型の教育からの脱皮 ===> データに基づいた問題解決力の育成
- 社会を生き抜いていくための有用なスキル
- [懸念] 高校の教員が対応できるのだろうか?
-
ビッグデータ時代のデータサイエンス
- 分析結果の質 <=== データの質に依存
-
「データの取り扱い」を中心に据えた分析姿勢
- 「膨大なデータを前にして本質を見抜く力」
- 重要: データ+教育+現場主義
9. Q3を終えるにあたって
初めてのリモート講義ということで、毎回薄氷を踏む思いでした。何とかここまで辿り着けたという気持ちですが、まずはお付き合い下さり、どうもありがとうございました。
この講義を通して、「統計」や「データ解析」と言う言葉に 多少なりとも親しみを持っていただけただろうか? RやRstudioの使い方に始まり、多変量解析と呼ばれる統計手法を幾つか紹介したが、数式自身よりもその手法の考え方や利用目的に重点をおいて 説明したつもりである。
大量の数値群から内在する構造を見つけることが「解析」であり「統計の面白味」でもあると思う。 そのためには、理論や目的を知っている必要があるのは勿論だが、対象とするデータの背景を知っておくことや、統計ソフトを“道具”として使いこなす必要もあろう。
今後、新聞や雑誌と言った生活では勿論のこと、研究やいろいろな場面で、種々の数値列に出会うことになると思うが、提示された数値にはどの様な意味(と意図)があり、どう理解して、個々人としてどうアクションを起すかの、判断の道具立ての一つとして統計学を活用してもらえれば幸いである。
なお、今後、もし統計に関して何か疑問に出会い、私に相談してみたいと思った際は、遠慮無くご連絡下さい。
皆さんのご期待にどこまで応えられたか心許無い部分もありますが、8回の講義、お付き合いくださり、どうもありがとうございました。お疲れさまでした。
88. 参考
このページで取り扱ったプログラムだけを抜き出して以下に列挙しておく。
# ディレクトリの移動。必須ではない。個々人の設定に応じて。
setwd("D:/home_sub3/R_Dir") # ホームディレクトリに移動(Set Working Directory)
## setwd("C:/home/R_Dir") # ホームディレクトリに移動(Set Working Directory)
getwd() # 現在のディレクトリ位置を表示
list.files() # ファイル・ディレクトリ一覧を表示
setwd("Food") # ディレクトリを移動
list.files() # ファイル・ディレクトリ一覧を表示
# データの読み込み 100品目の食品データ
food<-read.csv("food.csv", skip=17,
header=TRUE, na.strings="NA")
dim(food)
colnames(food)
food[1:5,]
corrFood = cor(food, use="complete.obs" ) # 相関行列を計算
corrFood
eigenFood<-eigen(corrFood)$values # 相関行列の固有値を計算
eigenFood
# 固有値の減少傾向を視覚的に捉えるためためにプロットしてみる
plot(eigenFood, type="b", main="Scree Plot",
xlab="NUM", ylab="Eigen Value")
abline(h=1, lty=3) # y = 1 の横線を追加
# 因子分析1
FAresultFood<-factanal(x=food, factors=3,
rotation="none", scores="Bartlett") # scores="regression")
# 分析結果要約
print(FAresultFood, cutoff=0)
# 各サンプルの変換点(因子得点, scores)を10ケース出力してみる。
FAresultFood$scores[1:10,]
# 番号で描画してみる。
plot(FAresultFood$scores[,1], FAresultFood$scores[,2], type="n") # マークを描かない
text(FAresultFood$scores[,1], FAresultFood$scores[,2], cex=.75) # その場所に番号を表示
abline(h=seq(-2,3,1), lty=3)
abline(v=seq(-3,2,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)
plot(FAresultFood$scores[,2], FAresultFood$scores[,3], type="n") # マークを描かない
text(FAresultFood$scores[,2], FAresultFood$scores[,3], cex=.75) # その場所に番号を表示
abline(h=seq(-2,4,1), lty=3)
abline(v=seq(-2,3,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)
plot(FAresultFood$scores[,3], FAresultFood$scores[,1], type="n") # マークを描かない
text(FAresultFood$scores[,3], FAresultFood$scores[,1], cex=.75) # その場所に番号を表示
abline(h=seq(-3,2,1), lty=3)
abline(v=seq(-2,4,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)
# 因子分析2
FAresultFood<-factanal(x=food, factors=3,
rotation="varimax", scores="Bartlett")
# 分析結果要約
print(FAresultFood, cutoff=0)
# 番号で描画してみる。
plot(FAresultFood$scores[,1], FAresultFood$scores[,2], type="n") # マークを描かない
text(FAresultFood$scores[,1], FAresultFood$scores[,2], cex=.75) # その場所に番号を表示
abline(h=seq(-2,3,1), lty=3)
abline(v=seq(-2,2,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)
plot(FAresultFood$scores[,2], FAresultFood$scores[,3], type="n") # マークを描かない
text(FAresultFood$scores[,2], FAresultFood$scores[,3], cex=.75) # その場所に番号を表示
abline(h=seq(-3,1,1), lty=3)
abline(v=seq(-2,3,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)
plot(FAresultFood$scores[,3], FAresultFood$scores[,1], type="n") # マークを描かない
text(FAresultFood$scores[,3], FAresultFood$scores[,1], cex=.75) # その場所に番号を表示
abline(h=seq(-2,2,1), lty=3)
abline(v=seq(-3,1,1), lty=3)
abline(h=0, lty=1)
abline(v=0, lty=1)